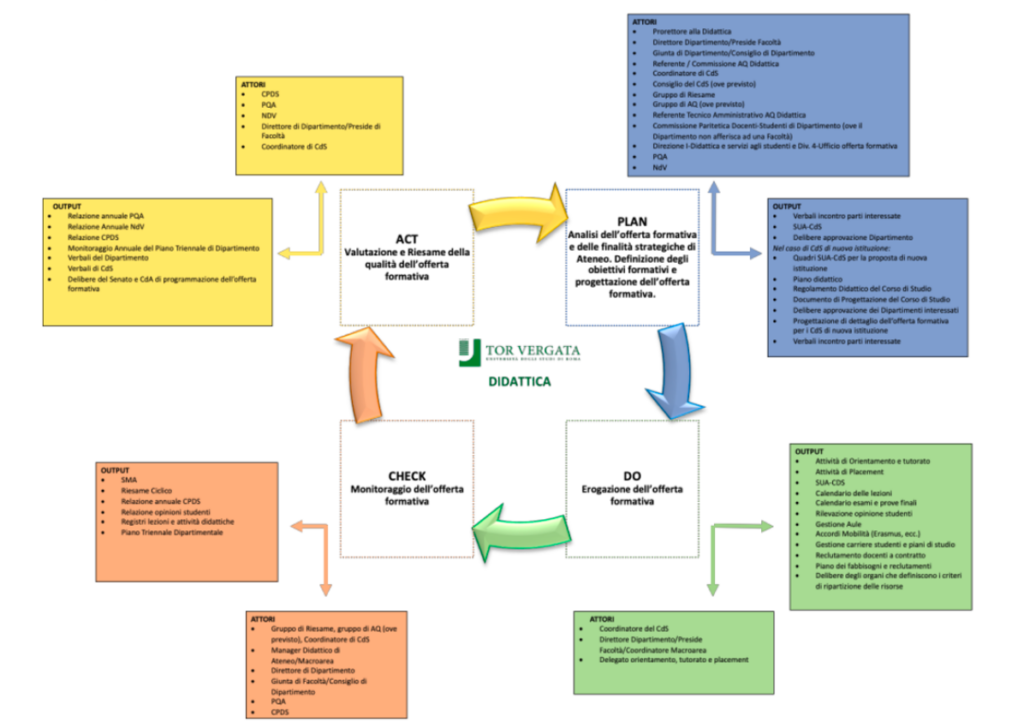
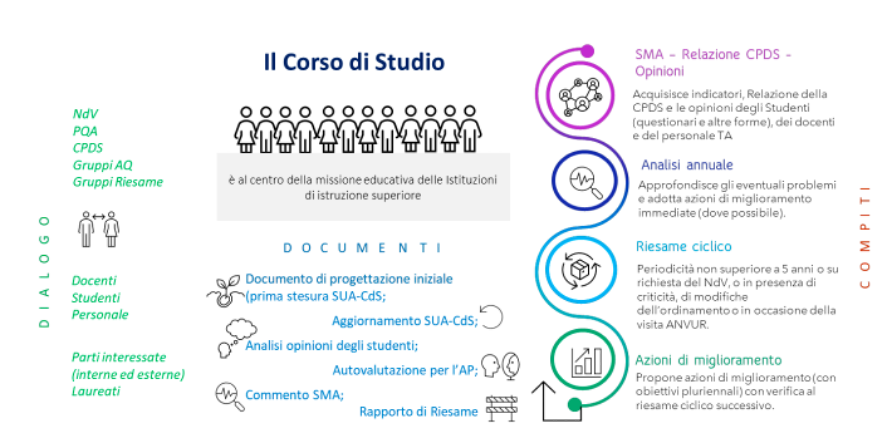
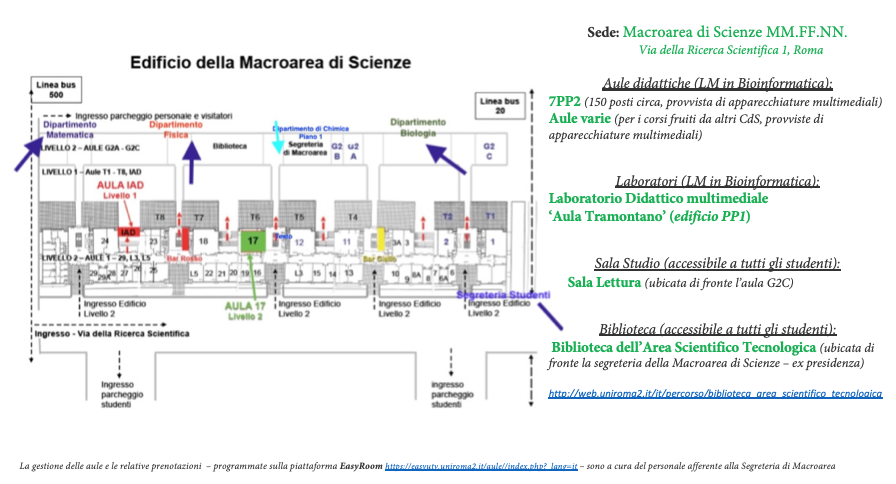
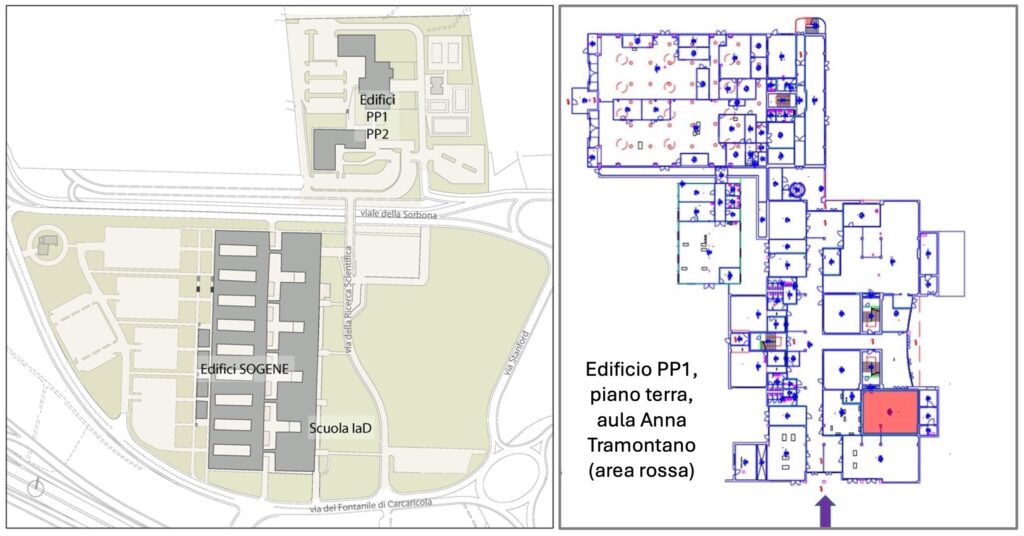
CdS Bioinformatica
- Sintesi dell’Incontro del 12 marzo 2021
- Sintesi dell’Incontro del 28 maggio 2019
- Sintesi dell’Incontro del 14 febbraio 2019
- Sintesi dell’Incontro del 22 ottobre 2018
- Sintesi dell’Incontro del 19 gennaio 2018
- Sintesi dell’Incontro del 25 settembre 2017
- Sintesi dell’Incontro del 01 agosto 2017
- Sintesi dell’Incontro del 06 aprile 2017
- Sintesi dell’Incontro del 16 dicembre 2016
- Sintesi dell’Incontro del 27 aprile 2016
- Sintesi dell’Incontro del 10 dicembre 2014
 UNIVERSITA' DEGLI STUDI ROMA TOR VERGATA
UNIVERSITA' DEGLI STUDI ROMA TOR VERGATA